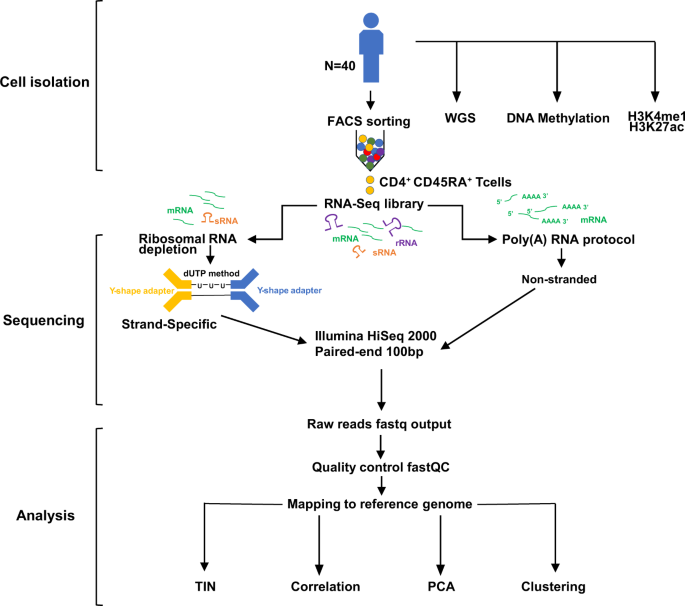
RNA-SeqによりRNA配列を解析したいと考えても、実際のRNAには様々な種類が存在し、異なる割合で存在します。特にヒトなどの真核生物では、rRNA(リボソームRNA)は全RNA中の80%以上存在するため、予めrRNAを除去しておかないと、mRNAなど他の目的とするRNAを十分に解析することが出来ません。
rRNAを除く代表的な手法として、サンプル中からrRNAを除く手法(rRNA depletion)と、Poly(A)を用いてmRNAを回収する手法(Poly(A) selection/enrichment)があり、本記事では各手法とその比較に関して解説します。
rRNA depletion
サンプル中からrRNAを除く手法です。Illumina様からRibo-Zero Kitの名称で試薬が販売されており、Ribo-Zeroの名前だけはお聞きしたことがある方もいらっしゃるかもしれません。Illumina様のRibo-Zero Kitは2018年に一度販売を終了しましたが、現在はrRNAを除去する機序を変え、Ribo-Zero Plusの名称で販売を行っております。
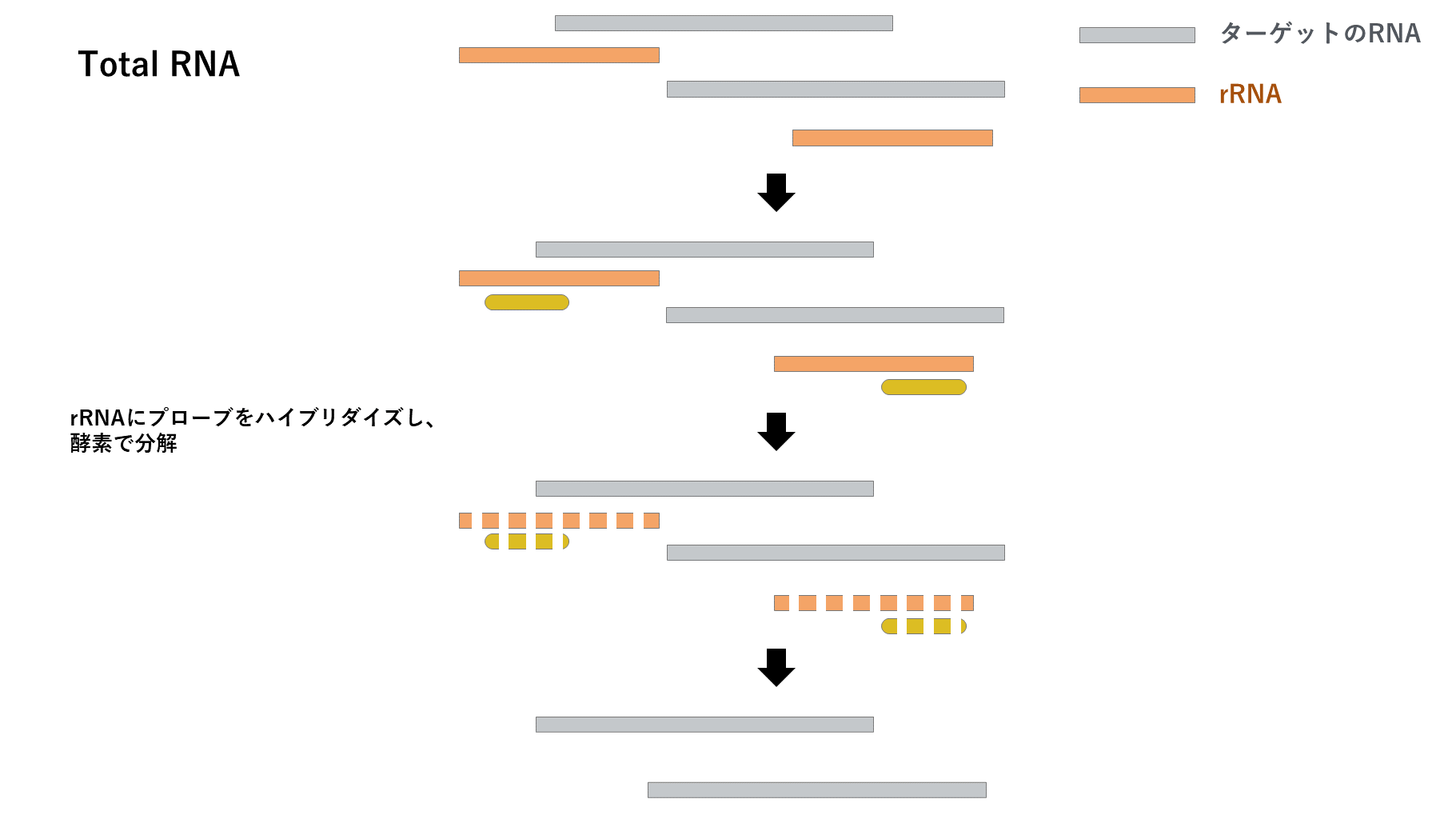
rRNA depletionは、(A) rRNAとビオチン付きプローブをハイブリダイズしマグネットにより除去する(Ribo-Zero)か、(B) rRNAとプローブをハイブリダイズし酵素で分解する手法(Ribo-Zero Plus)が主流です。
(A) rRNAとビオチン付きプローブをハイブリダイズしマグネットにより除去(Ribo-Zero)

(B) rRNAとプローブをハイブリダイズし酵素で分解(Ribo-Zero Plus)

Poly(A) selection/enrichment
多くの真核生物はmRNAの3’末端にAが連続する配列(Poly(A))を持っているため、相補的なTが連続する配列のビーズ(Oligo(dT)ビーズ)を用いて、濃縮することが可能です。
– Poly(A)を持つmRNAにOligo(dT)ビーズをハイブリダイズし、マグネットで濃縮

rRNA depletionとPoly(A) selection/enrichmentの比較
- 対応可能なサンプル
rRNA depletion法の場合は生物種に関わらず使用できますが、poly A selection/enrichment法の場合は上述したように真核生物が持つPoly(A)配列が必要になるので、Poly(A)配列を持たない細菌などの原核生物では利用できない方法になります。
- 解析できるRNAの種類
上記の図では省力しましたが、Total RNAにはrRNAやmRNA以外にも、non-cording RNAやtRNAなどの多様な種類のRNAが存在しています。

rRNA depletion法の場合はrRNAのみを除去するため、他の種類のRNAはサンプル中に含まれ、解析することが可能です。
対してPoly(A) selection/enrichment法の場合はmRNAのみを濃縮するため、他の種類のRNAは解析することが出来ません。ただし遺伝子発現解析を目的とする場合は、mRNAのみをターゲットすれば良いため、余計な種類のRNAを解析しないことでコストや解析の手間を減らすことが出来ます。
- 劣化したサンプルへの対応
Poly(A) selection/enrichment法の場合は3’末端のPoly(A)配列から濃縮するため、サンプルが劣化しており配列が短くなっていると、mRNAの5’末端側の配列は取得できなくなってしまいます。サンプルの品質に不安がある場合は、ターゲットがmRNAのみの場合でも、他の種類のRNA分データ量が必要になりますが、RNA depletion法を選択した方が良い場合があります。
本記事ではRNA-SeqにおけるrRNAの除去方法に関して、ベーシックな点を紹介しましたが、全血サンプルを扱う際はグロビン mRNA除去も検討が必要になるなど、未だご紹介できていない注意点があります。今後の記事で別途ご紹介できればと思いますが、RNA-Seqの前処理に関しては、下記の比較論文等ある程度の知見が集まっている点もあります。実際に実験内容が決まってきましたら、本記事の基本情報を基に、論文や各メーカーの試薬サイト等をご確認頂くことをお勧めします。