次世代シーケンス(NGS)に関する用語集。日々更新しております。
一覧のPDFファイルはページ最下部にご用意しております。
並びはアルファベット順→あいうえお順→数字順です。
サンプル配列(insert)の両端に付加するオリゴDNA。これらの配列を基にNGS機器でシーケンスを行う。

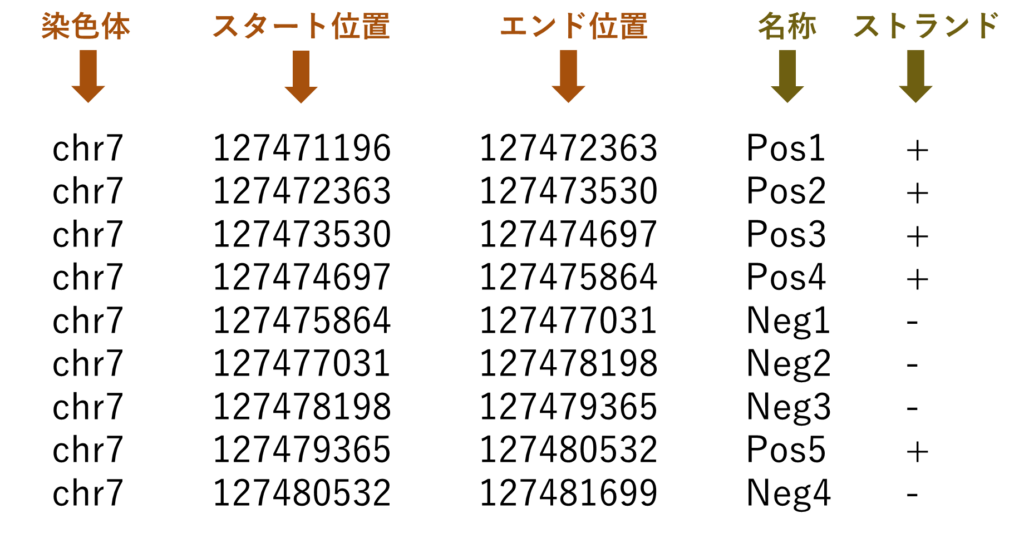
BED (Browser Extensible Data)。ゲノム上の位置を示すのに使われているフォーマット。染色体名、スタート位置、エンド位置は必須で、他にも任意で名称やストランド情報なども記載できる。
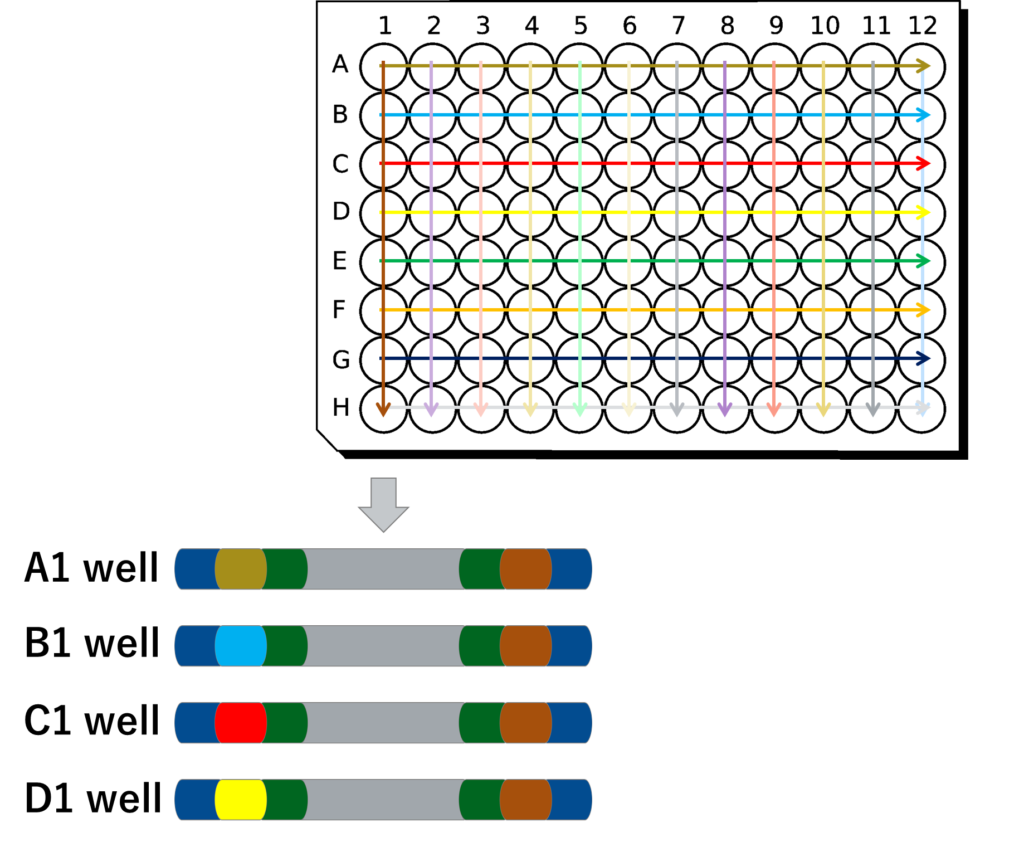
Dual Indexのひとつで、i5とi7の組み合わせで振り分け可能なサンプル数を増やす。index primerのコストを抑えながら、indexの組み合わせ数を増やすことができるが、index hoppingの問題があり徐々にUnique Dual Index(UDI)のユーザーが増えている傾向。
特定のサンプル配列に対して、シーケンス機器で読まれた数。10回読まれると10Xや10xのように表記される。NGSではPCRエラーやシーケンスエラーが発生するため、カバレッジ数が多いほど正確な結果とされるが、過剰に行うと無駄なコストが発生するため実験目的にあわせたカバレッジを計画する必要がある。
両側にインデックス配列をもつ。

ヒトゲノムのタンパク質をコードするエクソン領域のみを解析する手法。エクソン領域は全ゲノムの数%以下だが、タンパク質に翻訳される領域であることから、疾患に関わる変異の多くがエクソン領域にあると考えられている。そのため全ゲノムシーケンスよりも格段に低コストでありながら、重要な配列を解析できる効率の良い手法とされる。
塩基配列またはアミノ酸配列を表記するフォーマット。1行目は”>”で始まり配列の名称やコメントなどを記載し、2行目に実際の配列を記載する。

シーケンスした塩基配列とそのクオリティスコアを表記するフォーマット。1行目は”@”で始まり配列のIDと任意でコメントなどを、2行目に実際の塩基配列を、3行目に”+”とIDなどを、4行目に2行目の配列と対応する配列のクオリティ値を記載する。クオリティ値には文字や記号などが表記されているが、ASCII(というもの)を使用しており、1文字に対して2桁の数値で処理できる。

ヒトゲノムのレファレンス配列で2009年にリリースされたもの。数字が大きいものほど新しいバージョンとなる。
ヒトゲノムのレファレンス配列で2013年にリリースされたもの。数字が大きいものほど新しいバージョンとなる。
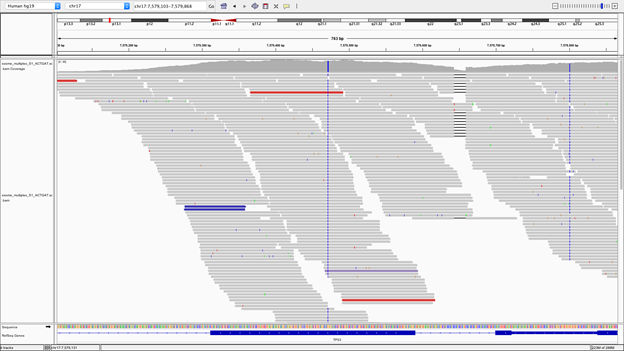
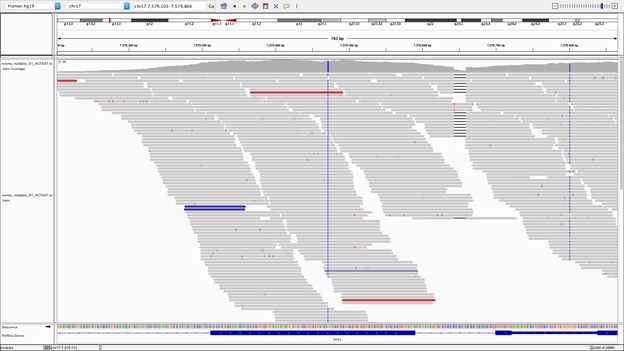
ゲノムブラウザの一つ。各ファイルデータを簡単に素早く視認できるため使用されることが多い。ソフトウェアをダウンロードしてローカルで使用できる。
IGVはIntegrative Genomics Viewerの略。
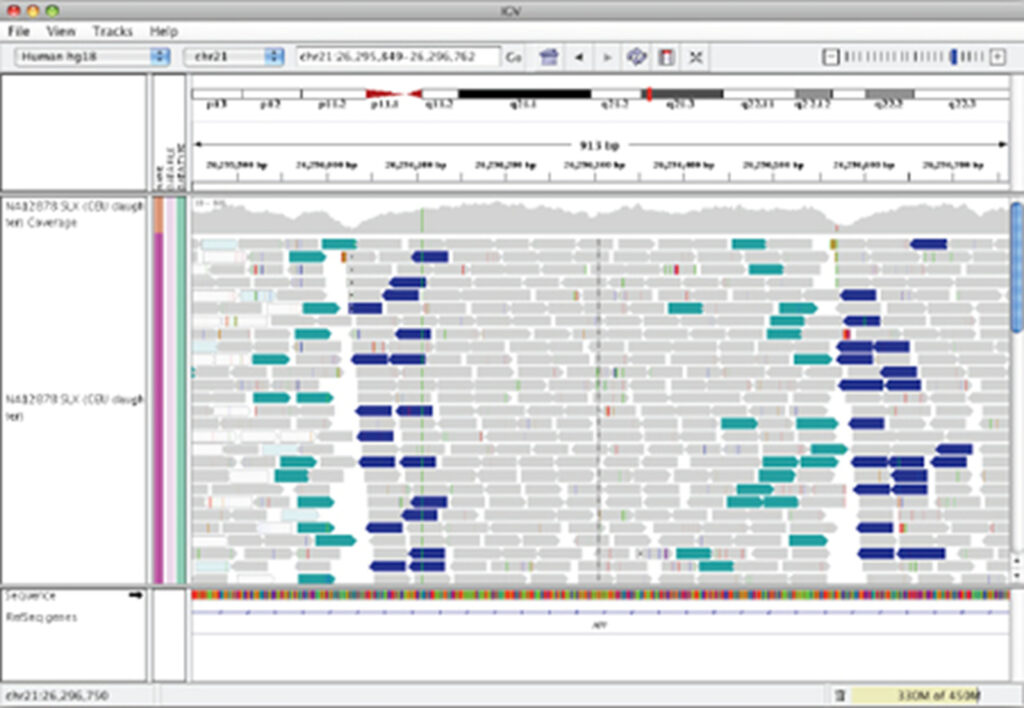
https://software.broadinstitute.org/software/igv/interpreting_pair_orientations
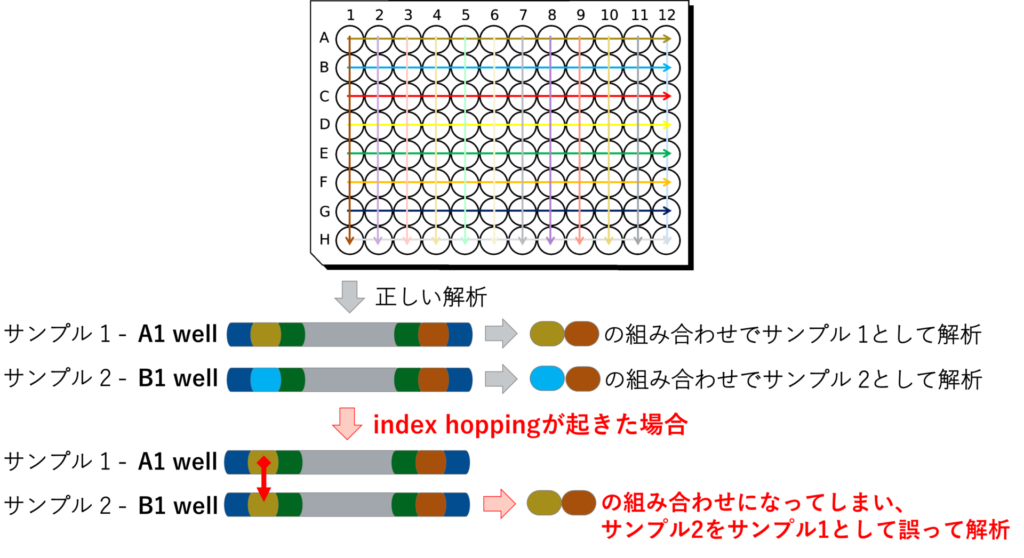
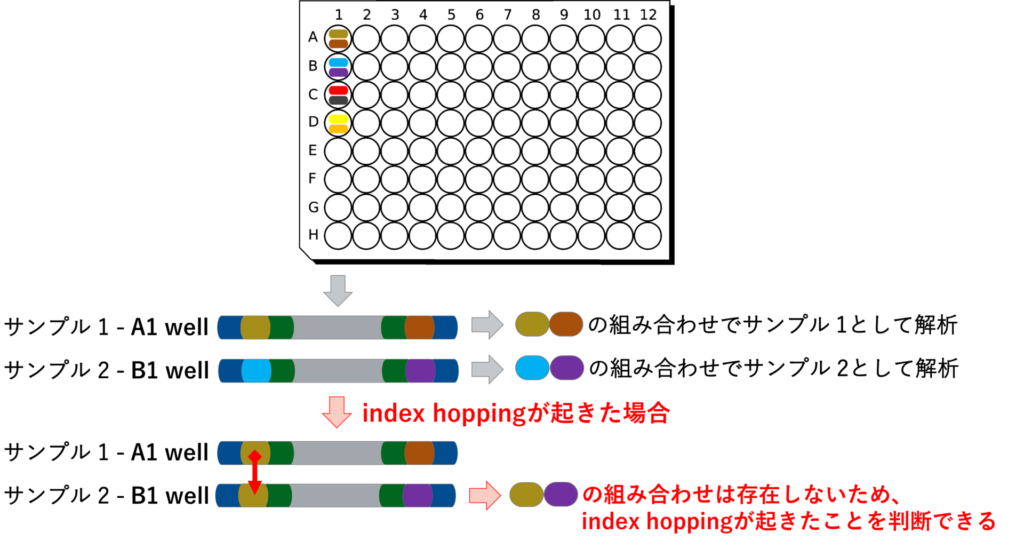
複数ライブラリーをプールした際にindexの組み換えが起こり、別のライブラリー由来のシーケンス結果がふくまれてしまう現象。Illuminaシーケンサーの、比較的新しい機種で採用されている方式(Patterned Flow Cell)で高い割合で発生したため、NGS業界では一時期大きな話題となった。
illuminaシーケンサーのインデックス/バーコード配列のこと。Dual Indexでは両端にインデックス/バーコード配列が付加されるため、i5とi7の名称で識別される。

マウスゲノムのレファレンス配列で2011年にリリースされたもの。数字が大きいものほど新しいバージョンとなる。
マウスゲノムのレファレンス配列で2020年にリリースされたもの。数字が大きいものほど新しいバージョンとなる。
ライブラリーの両側からシーケンスを読む。データ量はシングルリードの2倍になる。シングルリードと比べてコストが高いが、シーケンス結果から得られる情報も多く、より正確な解析が可能。
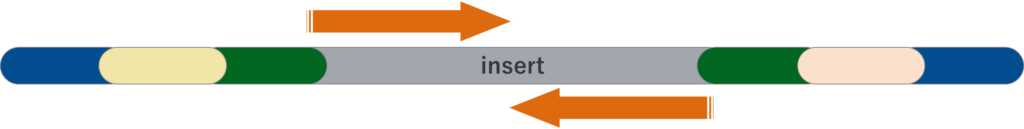
Illuminaシーケンサーのフローセルに結合するための配列。両端で異なる配列を付加する必要があるため、P5とP7の名称で識別される。

RNAサンプルをシーケンス解析する手法。DNAと違い転写物の発現量や、転写時の変異や融合遺伝子を検出することが可能。
片側のみインデックス配列をもつ。

ライブラリーの片側のみからシーケンスを読む。ペアエンドと比べてコストが低い。

オンラインでゲノムの各情報を確認可能なブラウザ。ヒトや主要のモデル生物に対応しており、各データベースにアクセスしたり、ダウンロードすることも可能。
名前の由来は開発した米国カリフォルニア大学サンタクルーズ校(UCSC:University of California, Santa Cruz)から。
ターゲット領域に対してどの程度均一なcoverageが取得できたかの指標。coverage uniformityとも言われる。特にターゲットシーケンスにおいてuniformityが高い(coverageが均一)なほど良いデータとされる。
Dual Indexのひとつで、i5とi7すべてで異なるindex配列を使用する。index hoppingの問題を大幅に低減することが可能。
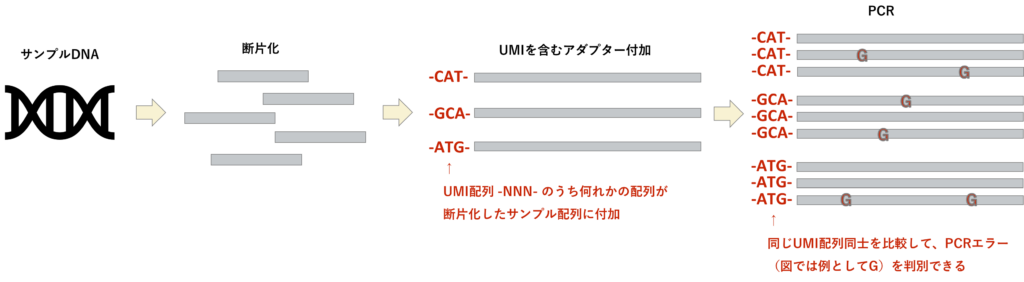
アダプターに含まれる、各サンプル断片に固有の配列。この配列を基に解析中にPCRエラーを判別でき、より精度の高い結果を取得できる。

対象サンプルの全ゲノム(すべての配列)をシーケンスする手法。対象サンプルの配列を漏れなく解析できるが、配列が多い分シーケンスや解析のコストが高い。10年ほど前まではヒトゲノム1人分1,000ドルが目標と言われていたが、現在目標はクリアし100ドルゲノムと言われることも。
ターゲットシーケンスの一つで、目的のサンプル配列のみをPCR増幅し解析する手法。PCRしながらライブラリー作製が出来るためプローブキャプチャー法より手間が少なく安価だが、プローブ設計自体が難しくPCRバイアスが発生しやすい。メタゲノム解析では、16S領域をアンプリコンシーケンスし、構成する菌種を特定していくことが多い。

ライブラリーの1本鎖DNAを基に、illuminaシーケンス機器で解析可能な状態まで増幅したもの。
サンプル中の特定の配列に絞ってシーケンスを行う手法。関心のある領域をより詳細に解析でき、余計な領域分にコストをかけず、解析の手間を減らすことができる。プローブキャプチャーまたはアンプリコンシーケンスによる手法が一般的。

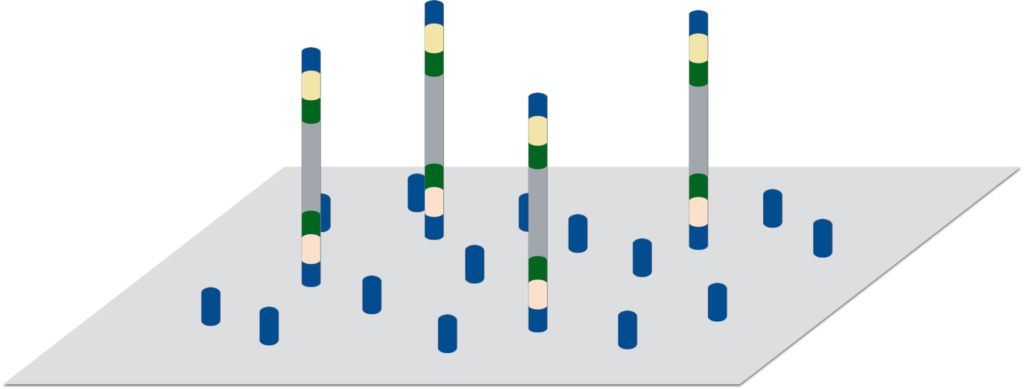
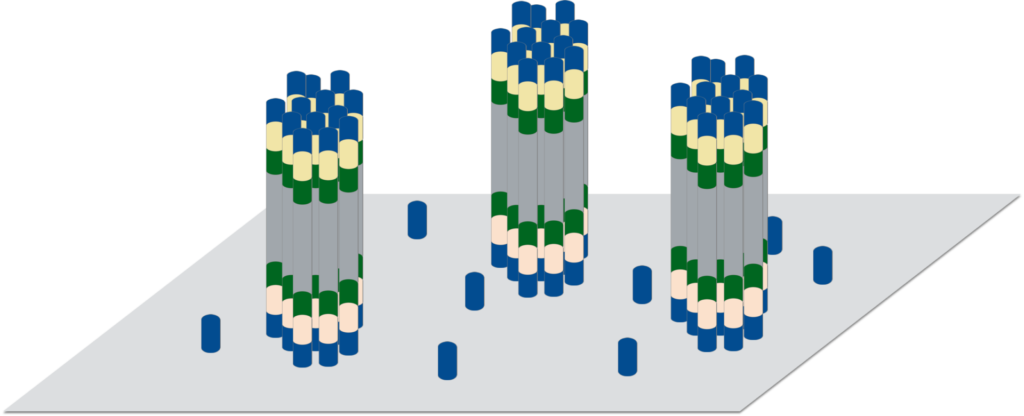
illuminaシーケンサーにおいて、P5/P7配列のオリゴが表面にあるスライドガラス。この表面に作製したライブラリーを流しクラスター形成後、シーケンスを行う。

ターゲットシーケンスを行う際に用いる、ターゲット特異的な配列のDNA/RNAオリゴ。ビオチン修飾が含まれており、ストレプトアビジンビーズと結合させて、目的の配列とハイブリダイゼーションした配列断片を濃縮する。

ターゲットシーケンスの一つで、目的のサンプル配列に相補的なプローブ/ベイトを用いて濃縮する。アンプリコンシーケンスよりPCRバイアスが少なく均一なカバレッジが取得出来るが、作業の手間が多くプローブコストが別途発生する。

サンプルDNAやRNAをシーケンス機器で解析可能な状態にしたもの。サンプルは適当な長さにし両側にアダプターが付加されている。

シーケンス機器によって読まれた各サンプルの配列。Illuminaでは正確だが読める長さが数百塩基と短いためショートリード、PacBioやNanoporeは数千~塩基読めるためロングリードと呼ばれる。
illuminaシーケンサーのフローセル上にある、ライブラリーを流す通り道。

平均カバレッジ(mean coverage)の0.2X以上となったターゲット配列の割合。主にアンプリコンシーケンス時の均一性の指標に利用される。例えば極端にカバレッジが低い領域があると、0.2X mean coverageの値が下がり均一性の低い結果と評価される。

