下記の記事でリード数とカバレッジの違いに関して簡単に説明しましたが、本記事ではNGS(次世代シーケンス)におけるカバレッジに関して、より詳細に解説します。
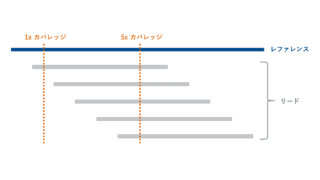
上記の記事でも解説しましたが、NGSにおけるカバレッジは、あるポジションや領域でリードが読まれた回数 = リードが重なる回数です。

NGSでは配列の読み取り精度は100.0%ではなく、エラーにより間違った配列が読み取られることがあります。その際に同じ配列を何度も読み比較することで、エラーの配列を認識できるようになります。そのためカバレッジが多い実験ほど、結果の精度が高いということになります。
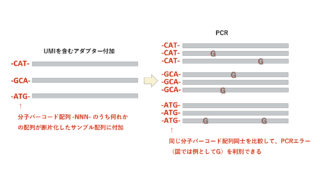
結果に必要十分とされるカバレッジは、実験目的によって異なります。例えば単にサンプルの配列を確認したいのであれば、30xやそれ以下でも良いかもしれません。もしサンプル中の変異を解析したい場合は、その頻度が低くく、より正確に検出したい場合ほど、より多くのカバレッジが必要になります。また分子バーコード(UMI)を使用した実験では、数万以上のカバレッジが必要な実験もあります。
カバレッジが多いほどより正確な結果となりますが、その分データ量が必要なりコストが高くなります。論文や過去の実績等から必要カバレッジを確認することが重要ですが、それでも手持ちのサンプルや実験内容によってカバレッジのバラつきもあるため、最初は余裕を持ったデータ量にすることも重要です。カバレッジが足りないと十分なデータを取得できず、実験やサンプルがすべて無駄になる恐れもあります。
カバレッジに必要なデータ量の計算方法は、基本的にはターゲットの長さ x 必要カバレッジで計算出来ます。例えばヒトゲノムは約30塩基長 = 3 Gb(ギガ塩基)なので、仮に配列を読むのに15xカバレッジが必要だとすると、3 Gb × 15x = 45 Gbのデータ量となります。実際はこのデータ量に加えて、duplicateなどの解析に使用しないデータや、PhiXも含まれますので、それらの値も考慮する必要があります。
PCRにより重複した配列で、通常は除外される。分子バーコードを用いた変異解析では、duplicateを残して変異かエラーか解析することもある。
バクテリオファージ。ゲノムサイズが小さく既知のため、サンプルに追加してシーケンスの評価に使用できる。またNGSラン時にサンプル配列のATGC割合が偏ると、シーケンスのクオリティーが下がるが、PhiXはATGCの多様性が高くサンプルに追加することで偏りを減らすことが出来る。
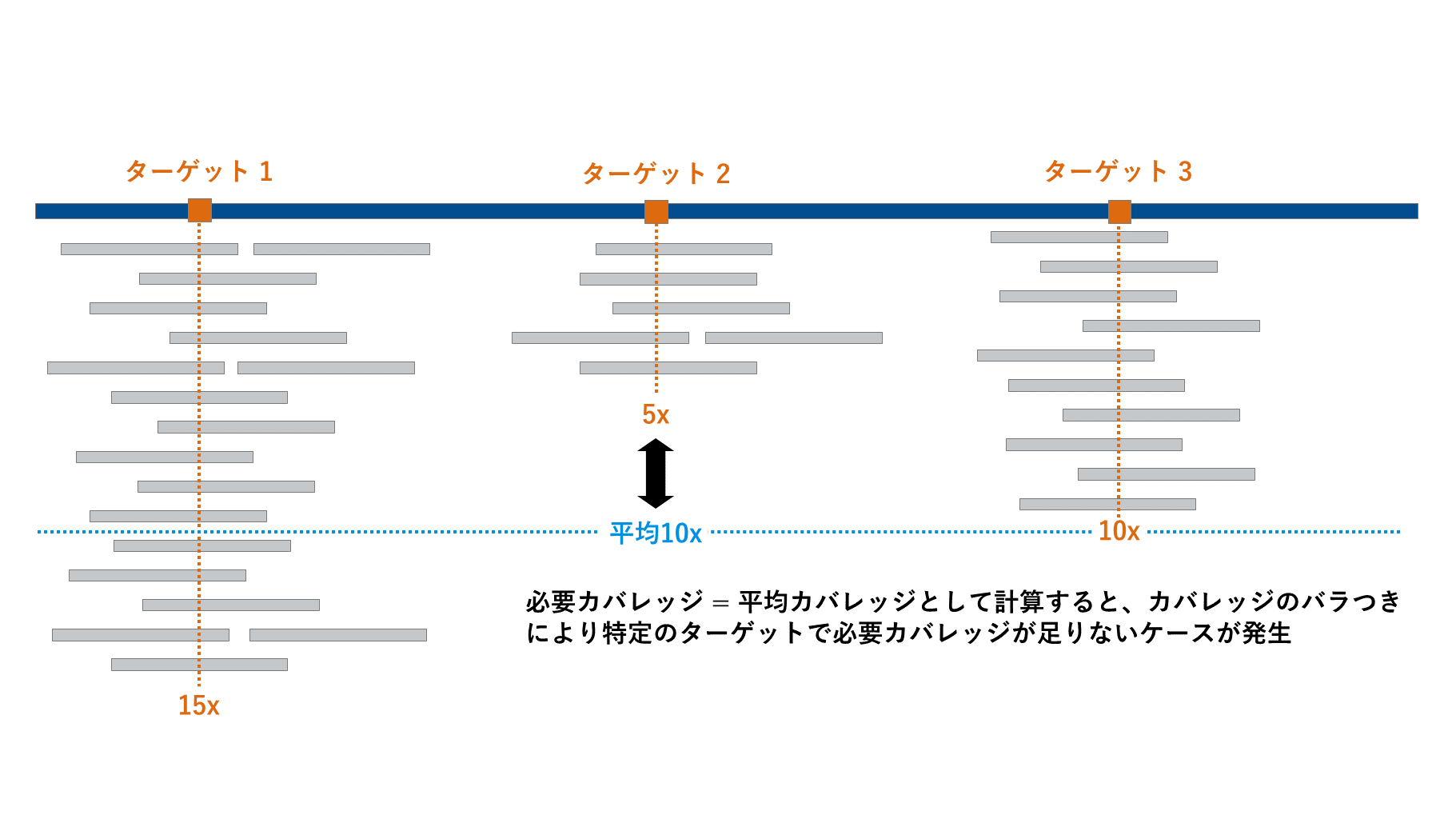
またターゲットシーケンスを行う場合は特に、各領域のカバレッジが均一にならない点に留意する必要があります。
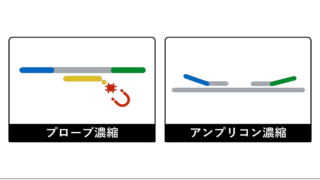
プローブキャプチャー法の場合は、ターゲット配列次第でキャプチャー効率に差が出ます。例えばターゲット変異の検出に10xカバレッジが必要と判断し、平均10xカバレッジのデータ量でシーケンスを行うと、バラつきにより解析したい変異箇所のカバレッジが少なく、データ量が足りないようなケースが発生するかもしれません。
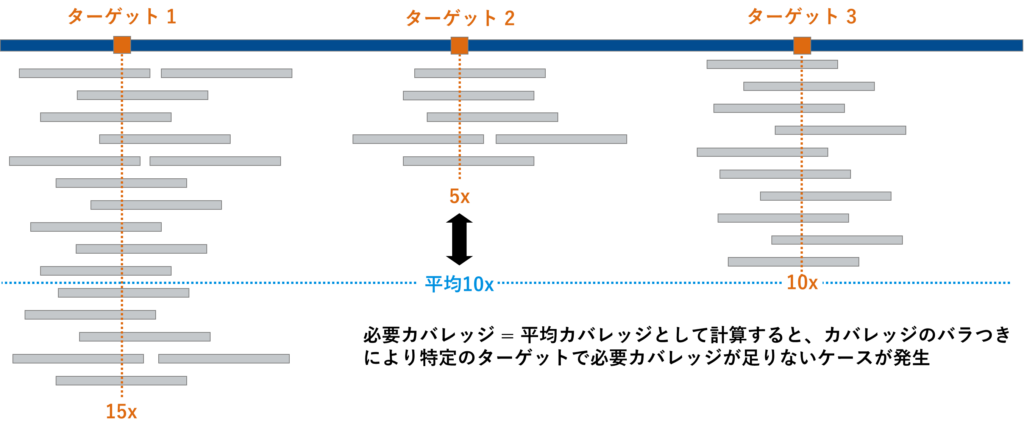
特にアンプリコンシーケンスはプローブキャプチャー法よりもカバレッジの均一性が低い傾向があり、必要なデータ量の設定に注意する必要があります。
GC含量が多い配列はカバレッジが低くなりやすい等の傾向はありますが、サンプルや実験内容次第な点もありますので、最初を余裕を持ったデータ量で確認することが大切です。