ライブラリー調製の際にシーケンスに必要なアダプター配列を付加しますが、Illuminaシーケンサーの場合、基本的には下記構造となっています。

実際のアダプター配列は別ページにご案内していますので、配列を確認したい場合は下記リンクからお進みください。
P5/P7
シーケンサーの基盤に結合する配列です。
i5/i7:インデックス配列
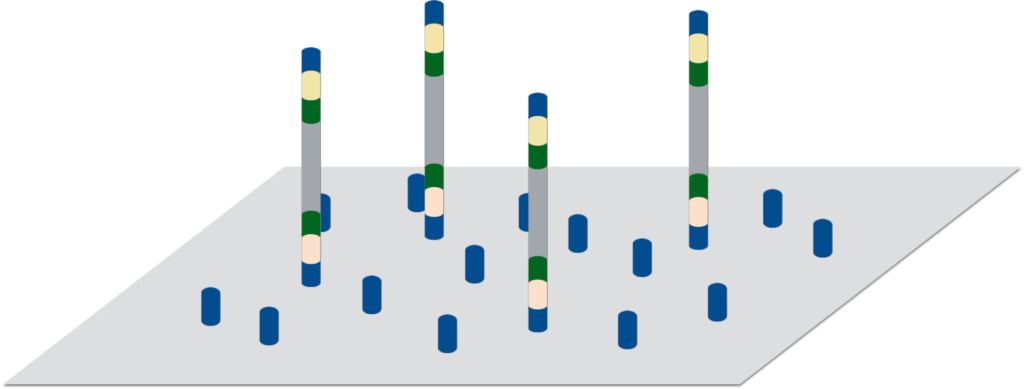
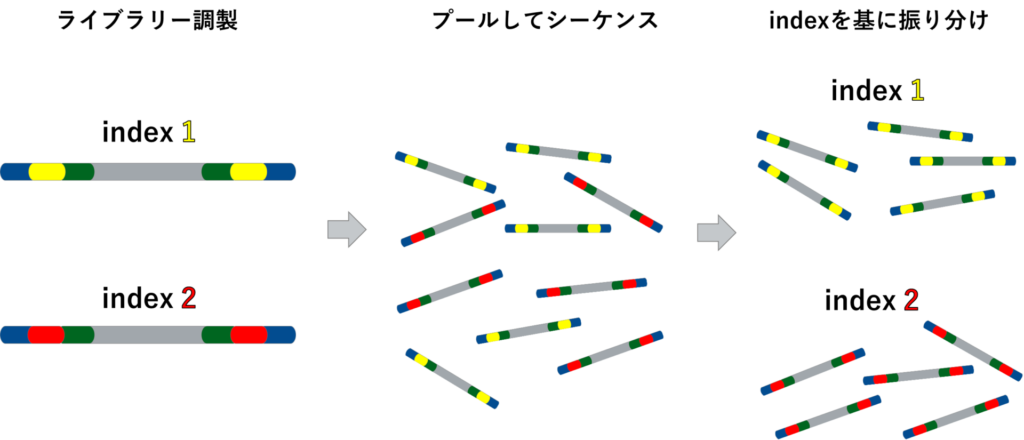
各サンプルに固有の配列です。シーケンス前に各サンプルをプールしても、インデックス配列を指標にデータを振り分けることが可能となります。複数サンプルをまとめてシーケンス出来るため、コストを抑えることが可能となります。
SP1/SP2:シーケンスプライマー配列
実際に配列を読み始めるプライマー配列です。下記図のようにRead1/2やRd1/2と表記されることも多いです。
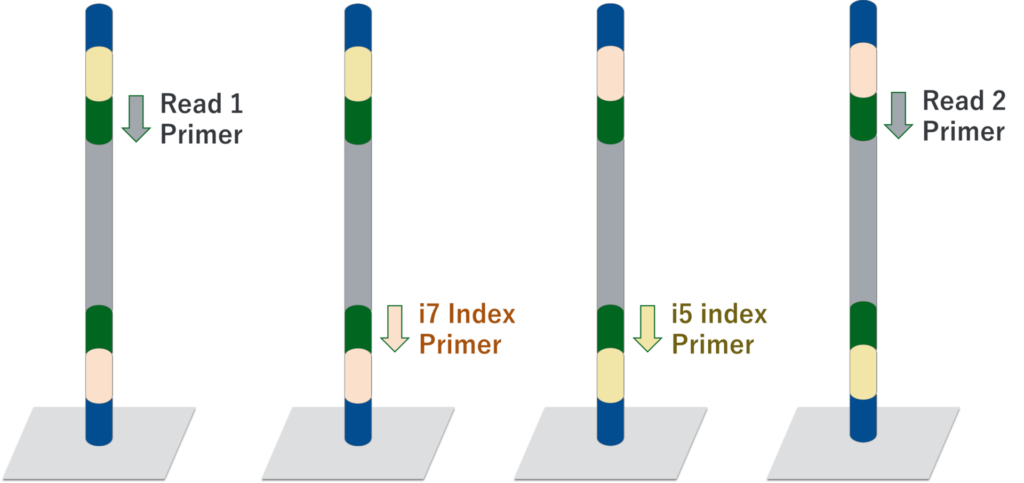
P5/P7とSP1/SP2はシーケンサーに必須で、指定の配列である必要があります。i5/i7はサンプルの振り分けを行わなければ除外してもシーケンスは可能ですが、シーケンス1ランで取得可能なデータ量と、1サンプル解析に必要なデータ量を考慮すると、多くのケースでindex配列は必要となります。
シーケンサーの開発段階でP1、P2、P3・・・と様々なプライマー配列をテストしたところ、P5とP7が最も良い結果となったようで、それ以来P5とP7の名前が採用されているようです。なおPはPrimer、iはindexの頭文字です。https://www.biostars.org/p/476156/
NGSに関する情報を調べていると、ライブラリーやアダプターがY型/枝分かれ・2本鎖・1本鎖といろいろなパターンで記載されていることがあり、NGSを実際に行ったことがない方は違いが気になるかもしれません。
厳密には2本鎖アダプターが直接付加されたライブラリーはY型/枝分かれ、PCR後のライブラリーが2本鎖になる等あります。ただ本記事のように概要を説明する場合は、本来はY型/枝分かれや別の形状になっているライブラリーも、模式的に1本鎖や2本鎖で表すことも多いです。
ちなみにIlluminaシーケンサーではシーケンスを行う直前で1本鎖にするため、どの形状のライブラリーで実験を行っても、シーケンス直前ではP5/P7 – インデックス配列 – シーケンスプライマー配列を持つ1本鎖になります。
・枝分かれ/Y型
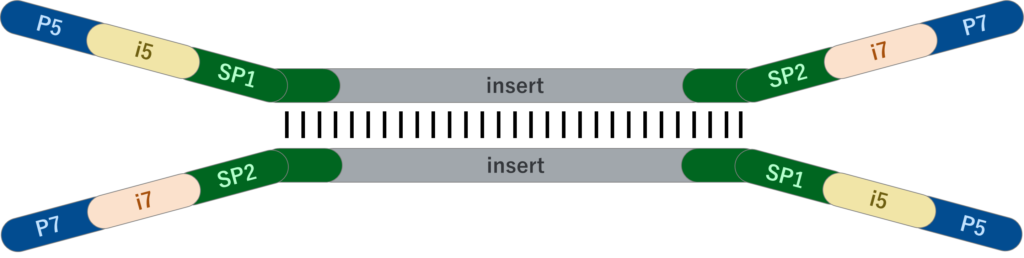
・2本鎖

・1本鎖





