IlluminaのNGSアダプターは最終的にP5/P7とシーケンスプライマー配列があればシーケンス可能となりますが、アダプターを完成させるまでの工程は様々です。各メーカーのキットを用いる場合や論文の手法に従って行う場合でも、その工程や違いを理解しておくと、手法の選択やトラブルシューティングに際に有用です。
本記事では数あるアダプター作製法のうち、比較的ベーシックな手法を幾つか説明します。
アダプターに必要な配列や、実際の配列は下記ページをご参照下さい。
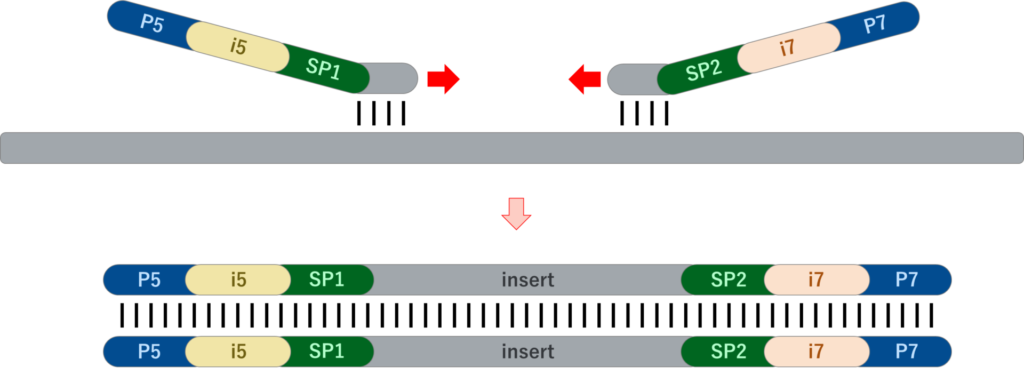
完全長アダプターのライゲーション
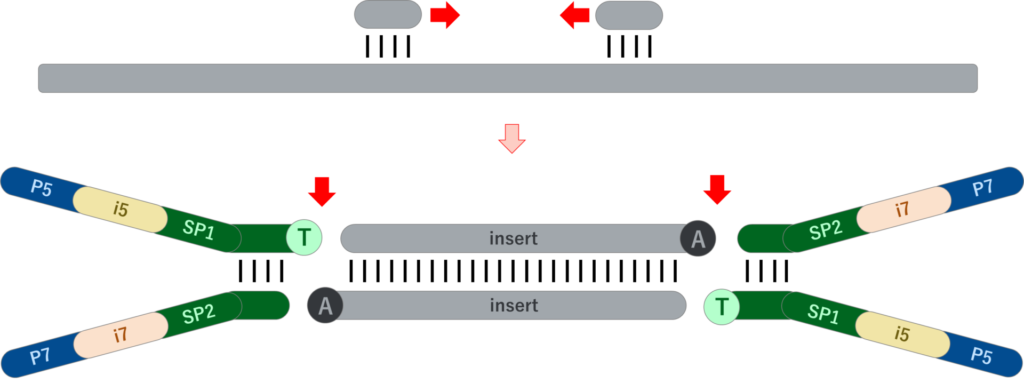
シーケンスに必要な配列(P5/P7 – インデックス – シーケンスプライマー)をすべて含む部分的2本鎖アダプターをサンプル配列(insert)にライゲーションする手法です。サンプル配列は両末端の片鎖にA塩基を付加する処理をしておき、アダプター側はT塩基が突出しているため、TAライゲーションにより結合します。
メリットとしては、アダプターを結合させるだけでライブラリーが完成するため、実験の工程が少なくて済みます。また後述する手法ではPCRが必要となりますが、この手法ですとPCR-freeでライブラリーが作製できるため、PCRバイアスが懸念される実験では第一選択となります。
デメリットは各インデックス配列ごと2本鎖アダプターを合成する必要があり、コストが比較的高価なことがあります。メーカーに2本鎖形成(アニーリング)を依頼すると、追加費用が発生し、納期も遅くなるかと思います。またサンプル配列にライゲーションするアダプターは、高い品質のオリゴが求められます。ただ以前よりは、各メーカーで提供している価格は安価になっている印象もあります。
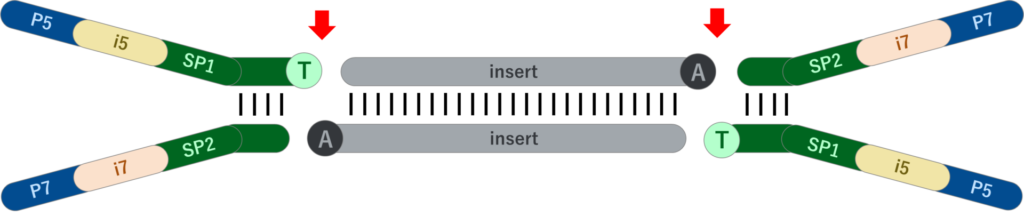
なお完全長アダプターのライゲーション後、P5/P7配列のプライマーにより、PCR増幅を行うことも可能です。
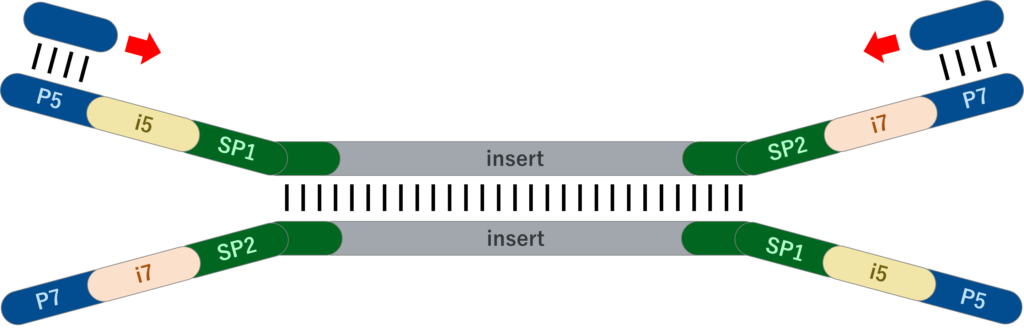
共通配列アダプターのライゲーション + インデックスプライマーのPCR付加
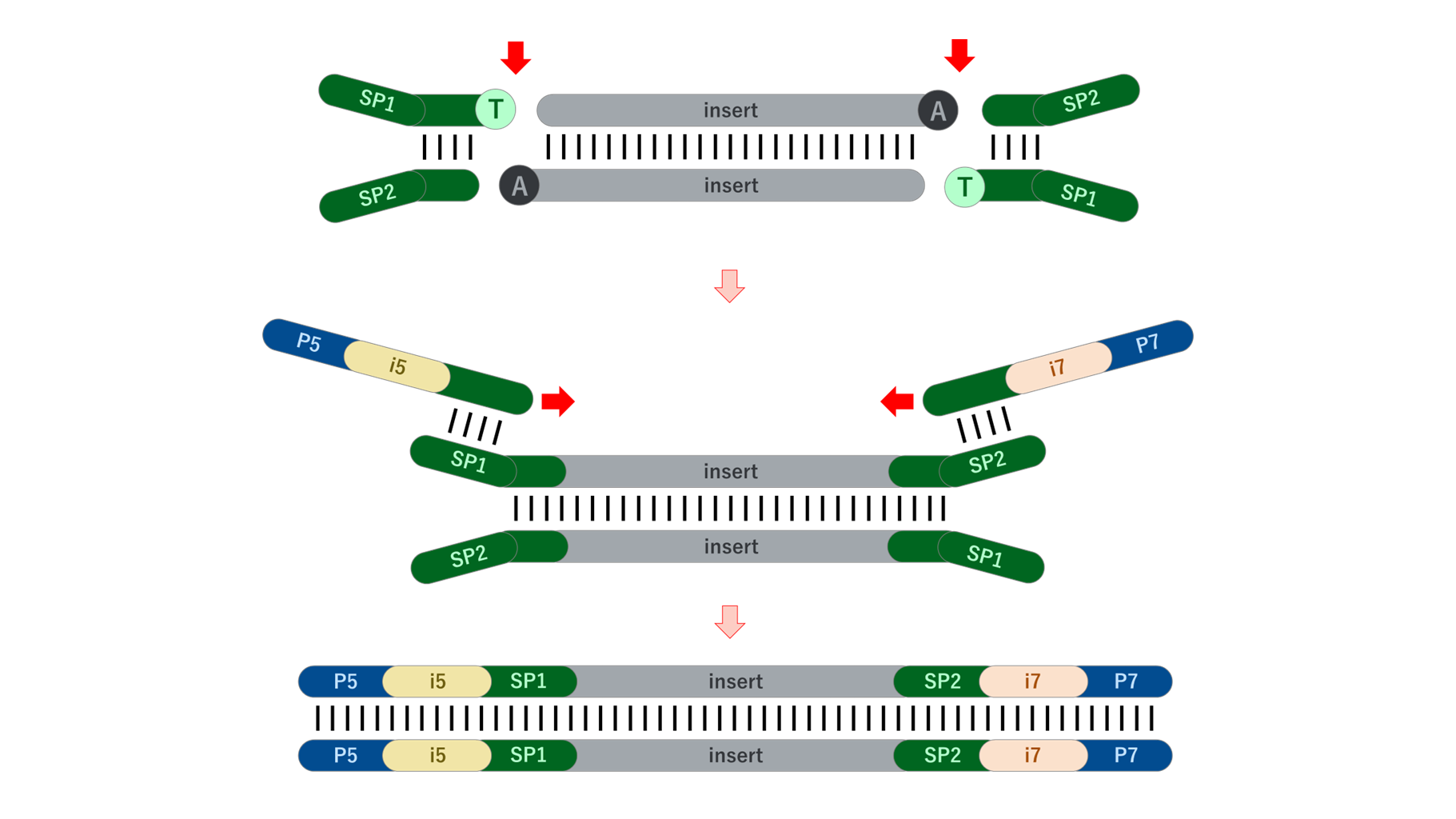
最初にシーケンスプライマーまでの共通配列を持つ、短い2本鎖アダプターをサンプル配列にライゲーションし、さらに各サンプルに固有のインデックス配列を持つプライマーをPCRにより付加する手法です。短い2本鎖アダプターは上記の完全長アダプターと同様に、TAライゲーションでサンプル配列に結合します。
メリットとしては、合成の手間が多い2本鎖アダプターを必要サンプル分 同じ配列で大容量合成すれば良い(= インデックス毎に2本鎖で合成する必要がない)ため、特にインデックス配列が多種類必要な場合、アダプター費用を完全長アダプターより安価に抑えることが出来ます。
例)96 サンプル同時にシーケンスしたい場合
→ 96 サンプル分の短い2本鎖アダプター(同じ配列) + 96 インデックス配列のプライマーペア(各サンプル分で固有の配列)
また2本鎖アダプターのライゲーションは配列が短いほど効率が良くなることが報告されており、完全長アダプターよりも効率よくライゲーションを行うことが可能です。
デメリットはPCRを行わないとライブラリーが完成しないため、PCR-freeの実験では使用できない手法です。
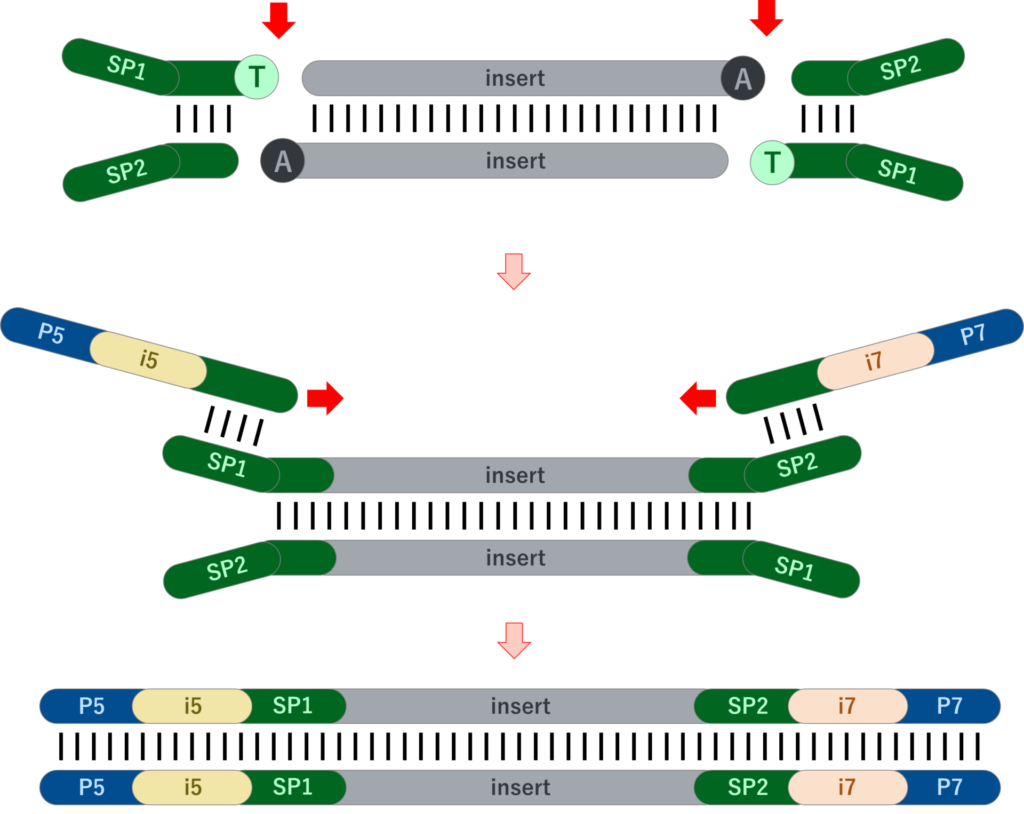
Nextera(Tagmentation)
Illumina様のNexteraキット独自の手法です。現在は名称が変更されNexteraは使わずTagmentationで統一されているようです。上記の2本鎖アダプターをライゲーションする手法では、ライゲーション前に超音波等の物理的な手法や酵素等により、目的にinsert長に断片化しておく必要があります。対してNextera(Tagmentation)のキットでは、transposomeにより断片化しながらシーケンスプライマーまでの配列を付加できます。その後はインデックスプライマーをPCR付加することで、ライブラリーを完成させます。
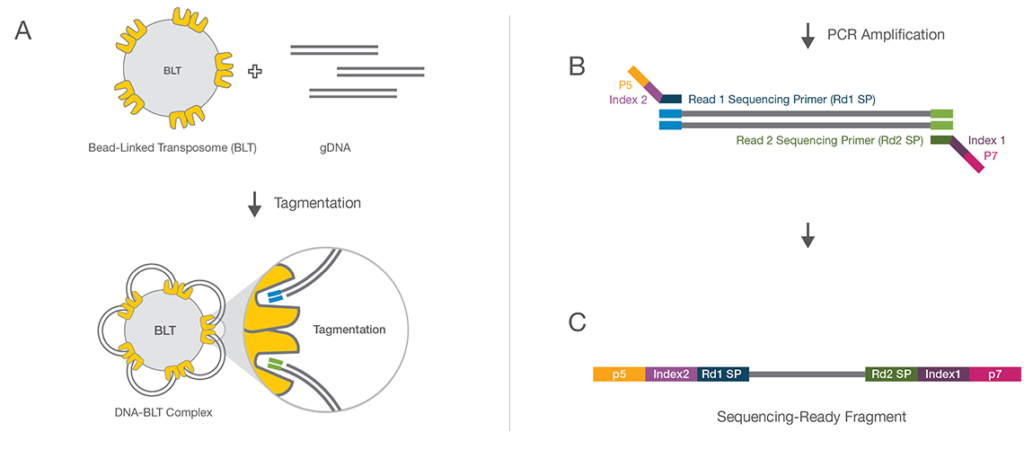
アンプリコン増幅 + インデックスプライマー
解析希望のターゲットをシーケンスプライマー配列を含むプライマーでPCR増幅し、その後インデックスプライマーをPCR付加することで、ライブラリーを完成させる方法させます。一般的にアンプリコンシーケンスと呼ばれる手法です。
2回のPCRのみでライブラリーを完成させることが出来るため、作業の工程が少なく済みます。ただし増幅したいターゲットが増えるほど、特異的なプライマー設計が難しくなりますので、カスタム設計の場合は実際の作業よりも設計が大変かもしれません。
また特定のターゲットを増幅する必要があるため、全ゲノム解析、プライマーを設計できない領域や設計しきれないほどターゲットが広い場合、またPCR-freeの実験では使用できません。
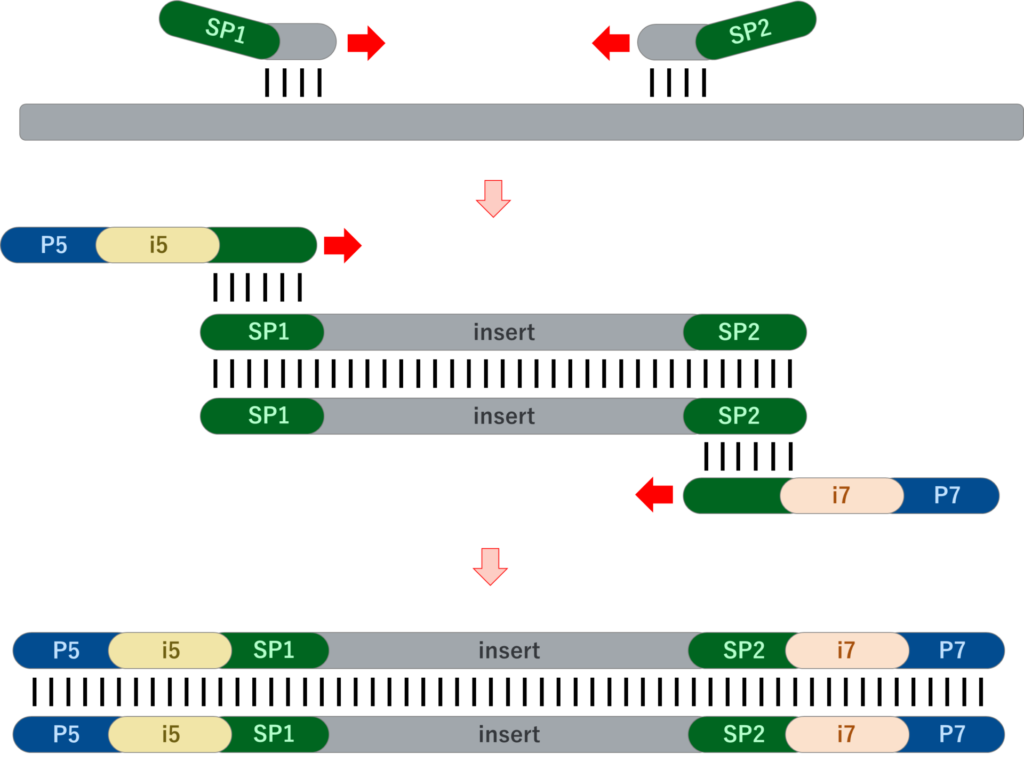
PCRを2回に分けず、アダプター配列とターゲット配列を含む長いプライマーを使用し、1回のPCRのみでライブラリーを完成させることも可能です。ただしターゲット配列 × インデックス配列の長いプライマーを用意する必要があり、プライマーの合成費用が高価になります。サンプル数が非常に多くプライマー合成コストよりも人件費や作業時間がネックになる場合や、ターゲットが少ない場合は、ご検討頂いて良いかと思います。
例)100箇所のターゲットを96 サンプル同時にシーケンスしたい場合
→ PCR 2回:100ターゲット分のプライマーペア(同じ配列) + 96 インデックス配列のプライマーペア(各サンプル分で固有の配列)
→ PCR 1回:100ターゲット分のプライマーペア(同じ配列) × 96 インデックス配列のプライマーペア(各サンプル分で固有の配列)= 9,600 プライマーペアを準備する必要あり
またシーケンスプライマー配列を含むプライマーを使用せずに、増幅したPCR産物にTAライゲーションを行いライブラリー完成させることも可能です。単にウイルス等の検出用に使用したプライマーとその増幅産物を、後に次世代シーケンスで配列を解析するといったことも出来ることになります。