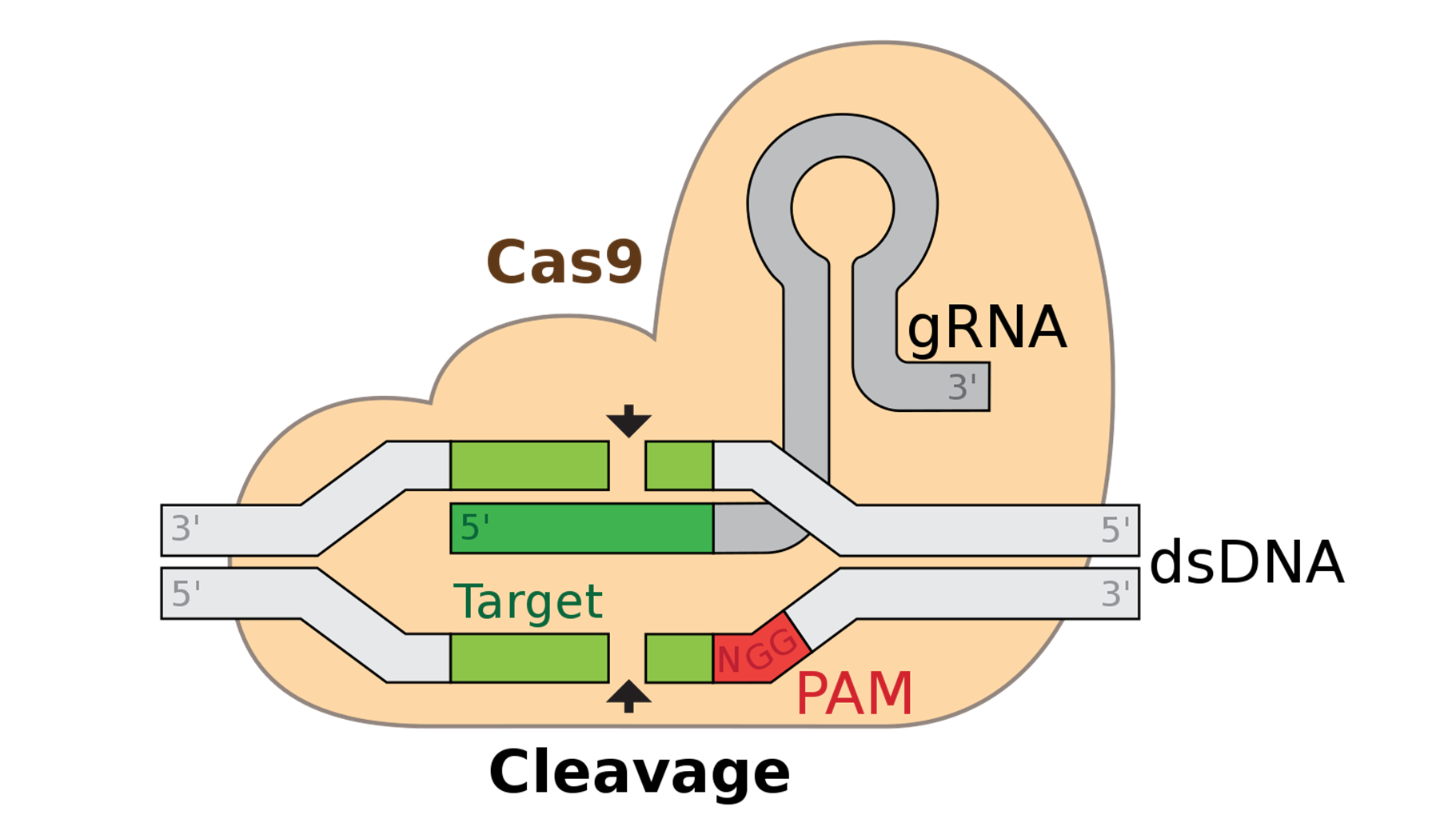
ゲノム編集とはDNA配列の特定の箇所を削除、追加、修正することで、目的とする遺伝子の働きをなくしたり、変えたりすることが可能な技術です。
ゲノム編集の技術自体は昔から存在していましたが、時間がかかる、配列が変わることが稀、ターゲットに出来る配列が限られる等の理由により、一般的な技術ではありませんでした。
それが2012年にCRISPR-Cas9が発表されると、上記の課題を大きく改善した手法として、世界中で飛躍的に利用されるようになりました。CRISPR-Cas9を発表した2人の研究者は、2020年にノーベル化学賞を受賞しています。
ゲノム編集技術の詳細はWikipediaなど他のサイトや書籍等で解説されてますので省力し、今回の記事ではCRISPR実験でNGS(次世代シーケンス)がどのように利用されているか解説します。

ゲノム編集実験で利用されるNGS
ゲノム編集、特にCRISPR-Cas9は簡単にDNA配列を変えることが出来ますが、幾つか課題もあります。その一つがオフターゲット(目的外の配列)への影響で、ゲノム編集実験を行った際は、オフターゲット配列に編集が起こっていないか評価する必要があります。
オフターゲットの確認方法は、特定領域のミスマッチのみを検出するキットから、各企業が開発したツールまで様々ありますが、網羅的に解析する手法としてNGSが利用されています。NGSを利用すると、オフターゲットが疑わしい領域をプライマーで増幅し網羅的に確認することも出来ますし、全ゲノムシーケンスによりサンプル配列全てを確認することも出来ます。
NGSを利用したゲノム編集実験の評価は、オフターゲット配列の確認に限らず、オンターゲット配列を含めたゲノム編集効率の評価にも広く利用されています。
特にNGSを利用した評価の利点として、定性と定量の両方の側面から網羅的な解析が可能な点があります。例えば特定領域のミスマッチのみを検出するキットでは、定性的な評価は出来るが定量的な評価は難しい場合がありますが、NGSでは配列取得による定性的な評価に加えて、リード数を比較することで定量的な評価も可能です。
もちろんNGSを利用する場合のデメリットもあり、実験の手間が多くコストがかかる点があります。特定の領域にゲノム編集が起こったかどうかのみを確認したい場合は、特定領域のミスマッチのみを検出するキットを用いた定性的な評価で十分な場合もあります。実験の段階にあわせて必要な評価方法を選択することで、効率よく経済的に実験を進めることが出来ます。
ゲノム編集技術を利用したNGS
ゲノム編集実験にNGSを利用したのではなく、ゲノム編集技術を利用したNGSとして、ターゲット領域両端のCRISPR-Cas9による切断およびシーケンスがあり、Oxford Nanopore Technologies社からはキット製品として販売されております。
NGSのターゲットシーケンスではアンプリコンシーケンス法またはプローブキャプチャー法で行うことが多いですが、CRISPR-Cas9によるターゲットシーケンスでは数 kb~程度のターゲット領域の両端を切断し、そのままアダプターを付加しロングリードのシーケンスを行います。
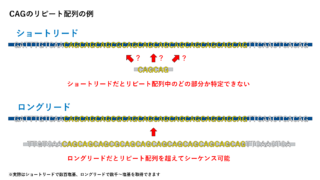
アンプリコンシーケンスでは長いフラグメントのPCRを行うことが難しいため、ロングリードを取得することが難しいのですが、CRISPR-Cas9によるターゲットシーケンスでは長いフラグメントを取得することが出来ます。またPCRを行わないため、ナノポアシーケンサーではメチル化なども修飾塩基も検出することが可能です。
またプローブキャプチャー法ではターゲットにリピート領域が多いと特異的なプローブ設計が出来ないのですが、CRISPR-Cas9によるターゲットシーケンスではリピート領域の両端に切断箇所を設計できれば、ロングリードシーケンサーの強みを活かしてリピート領域もシーケンスすることが可能です。
デメリットとしては、PCRを行わずにサンプル中の特定の領域しかシーケンスしないため、サンプル量を比較的多く準備する必要があります。