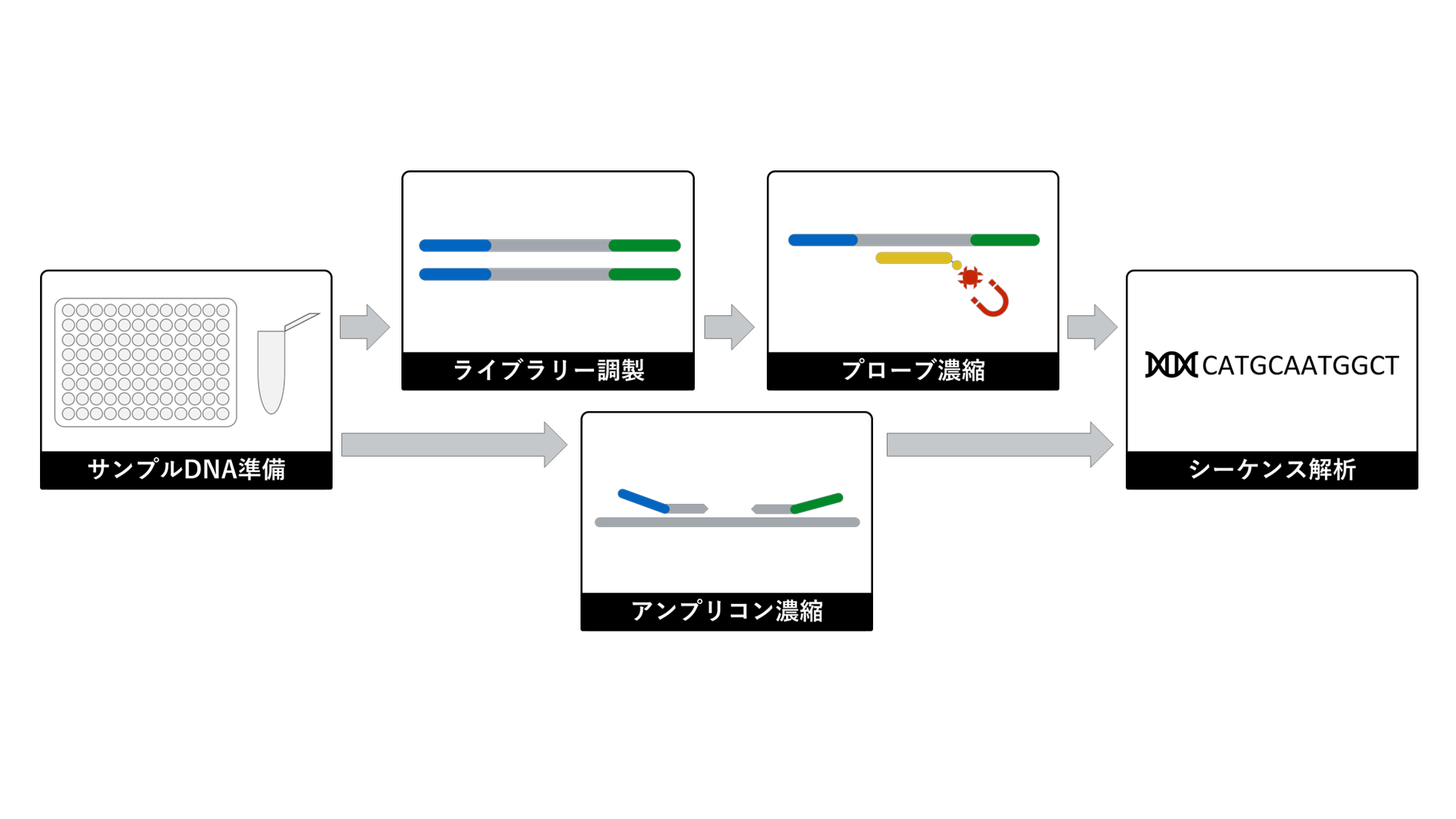
NGS実験では、DNAをそのままシーケンサーで解析できるわけではなく、いくつか前処理が必要になります。今回はIllumina機器の場合を例に記載しますが、他のシーケンサーも大まかな流れは変わりません。
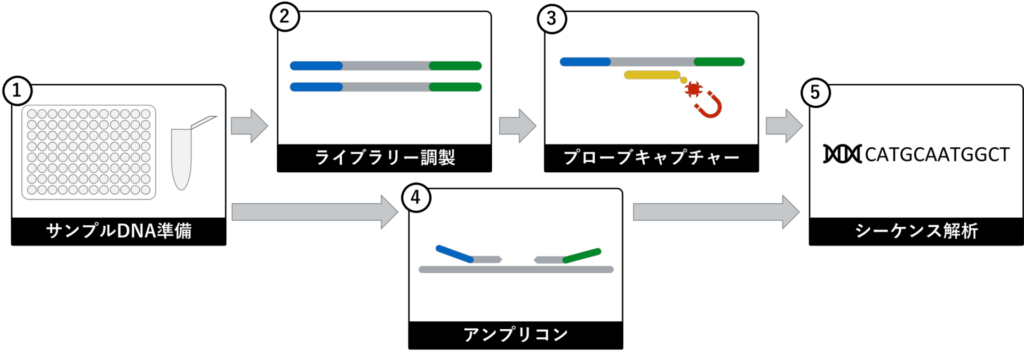
サンプルDNA準備
最初に解析したいサンプル DNA を準備します。NGS実験の解析対象は体 の血液や各組織から、腸内細菌など、解析対象は各種広がっているような状況にあります。 サンプルDNAは量が多く、品質が良いほど良好なシーケンス結果になります。逆に言えば、シーケンスに十分な量と質のサンプルDNAを準備できない場合、続く実験すべてが無駄になることもあります。
ライブラリー調製
サンプルDNAは、そのままの状態でシーケンスが出来る訳ではありません。DNAを適当な長さに切断し、各DNA断片にアダプターというオリゴをつけて、シーケンサーで解析できる状態にする必 要があります。シーケンサーで解析可能な状態にしたサンプルをライブラリーと呼びますので、このステップはライブラリー作製やライブラリー調製と呼ばれます。
プローブキャプチャー
ヒトゲノムは約30億塩基程度の長さになります が、実際に解析したい配列の領域はゲノム全体ではない場合があります。現状ではシーケンサーの 設定により決まった配列のみを解析するということができませんので、特定の領域のみを解析したい場合は、シーケンサーに持ち込む前にサンプルの配列を絞っておく必要があります。標的配列に特異的な修飾プローブを用いることで、目的の配列を含むライブラリーのみを濃縮することが可能です。
もちろん実験として、ゲノム全体を解析したいという場合もありますので、その場合はこのターゲット濃縮の工程をスキップすることも可能です。
アンプリコン
ライブラリー調製 – プローブキャプチャーの流れで目的の配列を絞ることも可能ですが、アンプリコン濃縮でも配列を絞ることが可能です。アンプリコン濃縮ではPCRにより目的の配列を増幅しながら、アダプターを付加していきます。アンプリコン濃縮はプローブキャプチャーと比べて実験工程が少ないのですが、PCRバイアスが多くなるなど、一長一短があります。
シーケンス解析
続いて準備したライブラリーや濃縮したライブラリーは、実際にシーケンスすることになります。下記の図では、あるサンプルを濃縮し、シーケンスした結果を示しています。
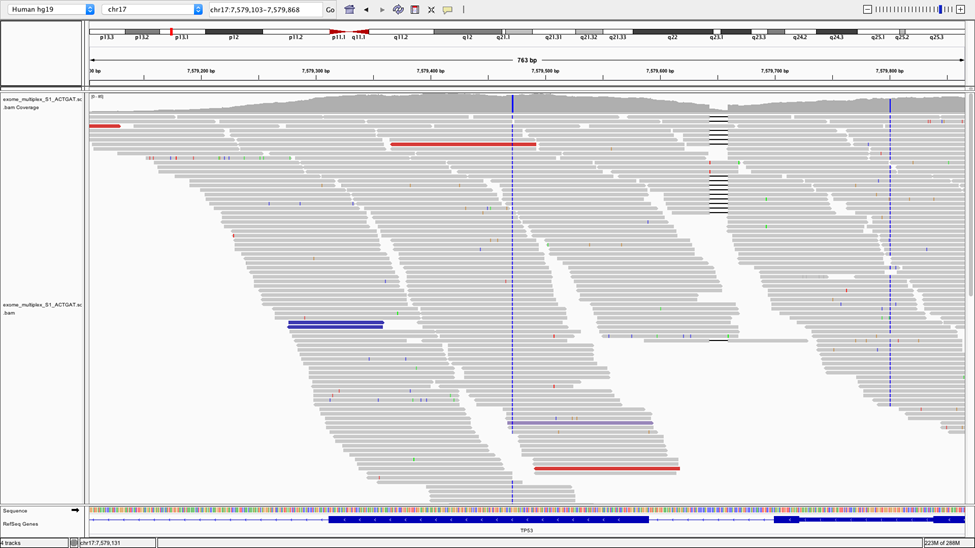
NGSによるシーケンスでは少なからずシーケンスエラーというものが発生いたしますので、希望の配列に対して異なる断片からシーケンスを行い、得られた配列を比較することでシーケンスエラーを見つけます。 そのためカバレッジと呼ばれる重なり合う配列が多いほど、結果の精度が高いことになります。このデータの場合ですと こちらの左端側の配列よりも、中心部分の配列の方が結果の精度が高いことになります。
特にサンプル中に存在する変異の割合が少ない場合などは、より高いカバレッジが必要になります。ただより高いカバレッジを取得するためには、そのぶんデータ量が必要になりますのでシーケンスコストも高くなります。