NGS実験において、シーケンス前のサンプルやライブラリーの品質評価(QC: Quality Check)は、良好な結果を取得しサンプルを無駄にしないために重要です。今回はNGS実験で使用される定性・定量方法に関して、ご紹介します。
Bioanalyzer/TapeStation
https://www.chem-agilent.com/contents.php?id=51
https://dornsife.usc.edu/uscgenomecore/bioanalyzer/
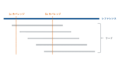
Agilent社さんが販売している機器で、チップにサンプルやライブラリーを充填することで、ゲル電気泳動を行うことが可能です。
チップや機器に多少コストはかかりますが、定性的/定量的な評価が出来るため、各社ライブラリー調製キットのプロトコールでもQCはBioanalyzer/TapeStationで行うと記載されています。またIllumina社さんはBioanalyzer結果を基にしたWebinarを行っており、トラブルシューティングの参考になります。
DNAに限らずRNAやタンパク質も測定でき、サンプル自体の品質、ライブラリー調製時の各工程、ライブラリー作製後と各段階でQCを行うことが可能です。
また近年では、FFPE等の分解が進んだサンプルから、シーケンスを行う需要が増えてきております。Agilent社さんはTapeStationの結果を基にした、独自のDNA Integrity Number (DIN)の指標を設定し、他社のキットでもDINを基にして実験条件を調整するなど利用されています。
https://www.agilent.com/cs/library/applications/low_5991-5258JAJP.pdf
qPCR
QCにおけるqPCRは、主に作製したライブラリーの定量評価に使用されます。Illuminaシーケンサーでは、適切なライブラリー量をロードすることが良好な結果を取得するために重要であり、量が正確に測定出来ていないとオーバークラスター等を発生させる恐れがあります。
またqPCRの場合は、アダプター配列に設計されたプライマーを基に測定するため、サンプル溶液中の正しく作製されたライブラリーのみを測定可能です。Bioanalyzer/TapeStationではサンプル溶液中のDNAを全て測定してしまうため、測定値と正しく作製されたライブラリー量が異なる恐れがあります。

Qubit
Thermo Fisher Scientific社さんが販売している、蛍光によりサンプルの定量を行う機器です。dsDNA、ssDNA、RNA、microRNA、small RNA、タンパク用のアッセイキットが販売されており、各サンプルを測定することが可能です。アッセイキットのスタンダードを基に、各サンプルを適宜希釈して測定します。測定結果は数値のみ表示されるため、例えば特定の配列長のdsDNAのみを測定したい場合でも、配列が長い/分解が進んだdsDNAも測定結果に含まれます。
NanoDrop
Thermo Fisher Scientific社さんが販売している、吸光度によりサンプルの定量を行う機器です。NGS実験では主にDNAの定量に使用されますが、RNAやタンパクの定量を行うことも可能です。希釈せずに簡便に測定を行うことが出来ますが、DNAと同じ吸光(260nm)をもつ物がコンタミしていると誤った値が測定されてしまい、今回紹介した手法の中では精度が低いと考えられるケースが多いです。実験手法が確立している、サンプル品質の差が少ない等、ルーティン的な作業で用いると良いようです。
まとめ
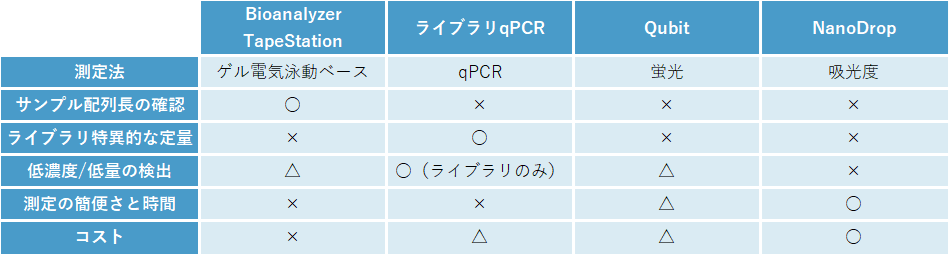
QC方法に関しては、各手法、装置、サンプルごとの違いを比較した、種々の論文が発表されています。それだけQCは、NGS実験を成功させるために重要な要素となります。
Quantification of massively parallel sequencing libraries – a comparative study of eight methods